不同參考基因組對RAD-seq分析中SNP?Calling的影響
隨著(zhù)新一代測序技術(shù)興起,基于酶切的簡(jiǎn)化基因組測序(Restriction-site associated DNA sequencing,RAD-seq)由于不受參考基因組的限制,成為快速獲取生物高密度單核苷酸多態(tài)性(single nucleotide polymorphism,SNP)的主流手段,被廣泛運用于變異檢測、遺傳圖譜構建、功能基因挖掘、群體進(jìn)化分析等研究中。盡管在RAD-seq分析中對SNP Calling方法的評估屢見(jiàn)不鮮,但在分析使用近緣種與物種本種作為參考基因組在SNP Calling上的差異關(guān)注不足,尤其是近年來(lái)隨著(zhù)全基因組測序物種的日益增加,深入探討不同參考基因組對SNP Calling的影響將為相關(guān)研究提供非常有價(jià)值的見(jiàn)解。
中國科學(xué)院西雙版納熱帶植物園(以下簡(jiǎn)稱(chēng)“版納植物園”)植物系統發(fā)育與多樣性保護研究組選取黃杞(Engelhardia roxburghiana)為研究物種,利用生物信息學(xué)軟件STACKS,探索使用不同參考基因組對SNP?Calling的影響。該研究圍繞兩種不同參考基因組:使用近緣種(即楓楊:Pterocarya stenoptera)作為參考基因組,以及利用物種本種(即黃杞)作為參考基因組。研究發(fā)現在使用物種本種作為參考基因組與使用近緣種作為參考基因組之間獲得的SNP數量存在顯著(zhù)差異,即本種作為參考基因組產(chǎn)生的SNP數量明顯多于使用近緣種作為參考基因組的情形。這一結果表明,選擇物種本種的參考基因組為SNP Calling的最優(yōu)方案;若物種本種參考基因組不可用,則可考慮使用近緣種的參考基因組,同時(shí)建議避免選用親緣關(guān)系較遠的物種。此項研究強調了參考基因組選擇對SNP Calling的影響,豐富了我們對RAD-seq數據分析的理解。
相關(guān)結果以Different reference genomes determine different results: Comparing SNP calling in RAD-seq of Engelhardia roxburghiana using different reference genomes為題,發(fā)表于Plant Science期刊。版納植物園植物系統發(fā)育與多樣性保護研究組2022級碩士研究生黃佩涵為該論文第一作者,孟宏虎副研究員、李捷研究員和上海辰山植物園的宋以剛副研究員為該論文通訊作者。該研究得到了國家自然科學(xué)基金、中國科學(xué)院東南亞中心項目、上海市園林綠化市容科學(xué)研究專(zhuān)項基金、中國科學(xué)院西部學(xué)者項目、云南省興滇英才支持計劃項目以及版納植物園“十四五”項目的支持。
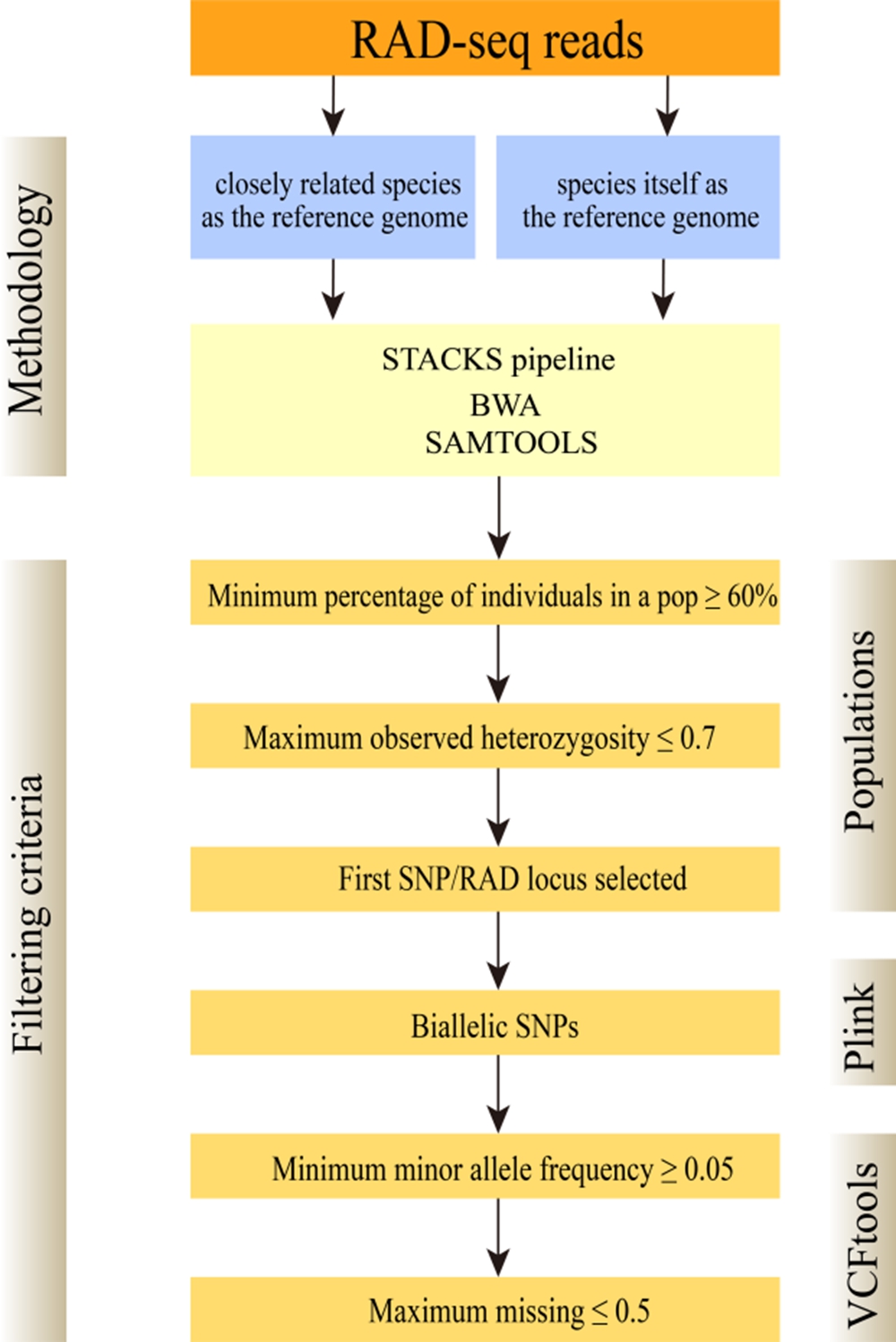
圖1研究分析方法流程
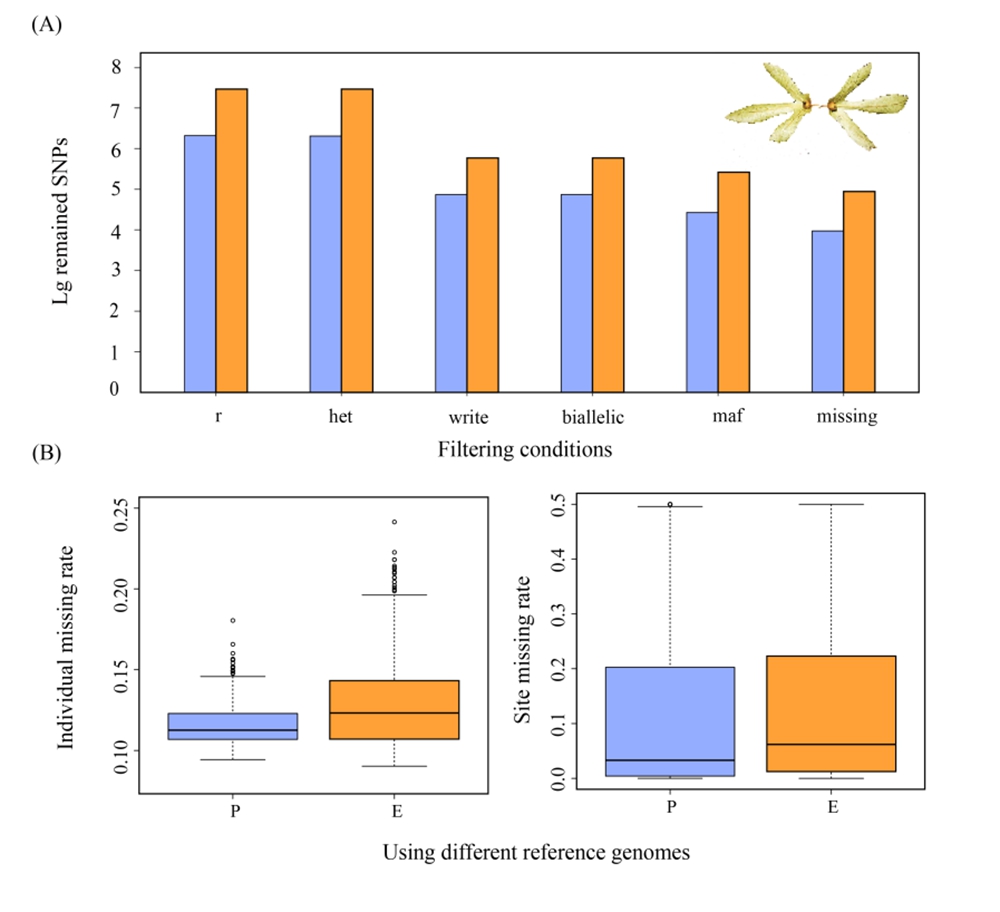
圖2?過(guò)濾步驟后保留SNP數量的結果,以及保留SNP的位點(diǎn)缺失率和個(gè)體缺失率。(A)不同參考基因組通過(guò)過(guò)濾步驟從初始到最終的SNP數量的lg轉換值。 (B)使用不同參考基因組,獲取的最終SNP的位點(diǎn)缺失率和個(gè)體缺失率(P:楓楊作為參考基因組;E:黃杞作為參考基因組)